Problème des molécules – Bac théorique – Section informatique – 2014

Sujet (Algo et programmation - Bac 2014)
Une molécule est un regroupement d'au moins deux atomes qui sont unis par des liens chimiques et elle est représentée par une formule chimique. Exemple : HO2.
Une formule chimique est une succession de symboles d'atomes, suivi chacun par un entier représentant le nombre d'apparitions (nbr) de l'atome dans la molécule.
Chaque atome est symbolisé par la première lettre de son nom en majuscule, suivie éventuellement d'une deuxième lettre en minuscule pour distinguer des atomes ayant des initiales identiques. Ainsi, le Fluor (F) se distingue du Fer (Fe), du Fermium (Fm) et du Francium (Fr),
Le calcul de la masse molaire moléculaire d'une molécule, notée NI(Molécule), sera comme suit :
- pour chaque atome de la molécule, calculer le produit (nbr x Matome)) ois A(atome) est un réel représentant la masse atomique de l'atome
- calculer la somme des produits obtenus.
Exemple :
Pour la molécule dichromate de potassium (K2Cr207) qui est constituée de 2 atomes de potassium. (K), 2 atomes de chrome (Cr) et .7 atomes d'oxygène (e), sa masse molaire moléculaire ill(K2Cr207) est égale à 2*.A(K) ± 2 *A ('Cr,) + 7 *.4 CO.
Puisque A(K) = 39,1 g/mol, A(Cr) = 52 g/ o1 et A(0) = 16 g/mol,
alors M(K2Cr207) sera égale à 2 * 39,1 + 2*52 + * 16 = 294,2 g/mol
En disposant d'un fichier texte "Molecules.txt" dont chaque ligne contient le nom d'une molécule suivi de sa formule chimique, séparés par le caractère astérisque "*", écrire un programme permettant de :
- remplir un fichier "Atomes.dat" par les données relatives à N atomes (N<50), où chacun est représenté par son symbole et sa masse atomique, - stocker dans un fichier "Resultats.dat" le nom et la masse molaire moléculaire de chaque molécule figurant dans le fichier "Molecules.txt". Travail demandé
1- Analyser le problème en le décomposant en modules.
2- Analyser chacun des modules envisagés.
Solution Algorithmique
Dans cet algorithme, On va utiliser six fonctions et quatre procédures:
- la fonction saisie()
- la fonction verif_atome()
- la fonction saisie_atome()
- la procédure remplir_fichier_atomes()
- la fonction verif_formule()
- la fonction saisie_formule()
- la procédure remplir_fichier_molecules()
- la procédure extraire_noms_nombres_atomes()
- la fonction recherche_atome()
- la procédure remplir_fichier_resultats()
Algorithme du programme Principal
|
1 2 3 4 5 6 7 8 9 10 11 |
Algorithme masses_molécules Debut longueur_noms <- 0 longueur_nombres <- 0 n = saisie(0, 51) remplir_fichier_atomes(n) m = saisie(0, 51) remplir_fichier_molecules(m) remplir_fichier_resultats(noms_atomes, nombres_atomes) Fin |
Déclaration des objets
| Objet | Type / Nature |
|---|---|
| longueur_noms | entier |
| longueur_nombres | entier |
| n | entier |
| m | entier |
| noms_atomes | tableau des chaînes |
| nombres_atomes | tableau des entiers |
La fonction saisie
Le rôle de cette fonction est de permettre à l’utilisateur de saisir un entier n compris strictement entre deux bornes inf et sup.
1- La fonction saisie(inf, sup) demande à l’utilisateur d’entrer un entier n.
2- Elle vérifie que n appartient à l’intervalle ouvert ] inf , sup [ (c’est-à-dire inf < n < sup). 3- Tant que la valeur saisie n’est pas valide (n ≤ inf ou n ≥ sup), la fonction redemande la saisie. 4- Une fois une valeur correcte entrée, la fonction retourne n.
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 |
Fonction saisie(inf:entier, sup:entier):entier # Première saisie de n Ecrire('donner n tel que ', inf , '< n < ' , sup, ': ')) Lire(n) # Tant que n n’appartient pas à l’intervalle ]inf , sup[ # on redemande la saisie Tant que (n <= inf) ou (n >= sup) faire Ecrire('donner n tel que ', inf , '< n < ' , sup, ': ')) Lire(n) Fin tant que # Retourne la valeur valide retourner n Fin |
Déclaration des objets
| Objet | Type / Nature |
|---|---|
| n | entier |
La fonction verif_atome
Le rôle de cette fonction est de vérifier si une chaîne de caractères représente un nom d’atome valide, c’est-à-dire composée uniquement de lettres.
1- La fonction verif_atome(ch) parcourt la chaîne caractère par caractère.
2- À chaque étape, elle vérifie si le caractère courant est une lettre de l’alphabet (majuscule ou minuscule).
3- La méthode Majus() permet de traiter les deux cas (A–Z et a–z).
4- La boucle s’arrête dès qu’un caractère non alphabétique est trouvé ou quand on atteint l’avant-dernier caractère.
5- Valeur retournée :
Vrai → la chaîne contient uniquement des lettres (nom d’atome valide)
Faux → la chaîne contient au moins un caractère non alphabétique (chiffre, symbole, espace…)
|
1 2 3 4 5 6 7 8 |
Fonction verif_atome(ch:chaine):booleen i <- 0 # Parcours de la chaîne tant que ce sont des lettres Tant que (i<long(ch)-1) et ('A'<= Majus(ch[i])<= 'Z') faire i <-i+1 Fin tant que retourner ('A'<= Majus(ch[i])<= 'Z') Fin |
Déclaration des objets
| Objet | Type / Nature |
|---|---|
| i | entier |
La fonction saisie_atome()
Le rôle de cette fonction est de permettre la saisie d’un nom d’atome valide et garantir qu’il est composé uniquement de lettres.
1- La fonction saisie_atome() demande à l’utilisateur d’entrer le nom d’un atome.
2- Elle utilise la fonction verif_atome(atome) pour vérifier la validité du nom saisi.
3- Tant que le nom contient des caractères non alphabétiques (chiffres, symboles, espaces…), la saisie est redemandée.
4- Une fois un nom valide entré, la fonction retourne ce nom.
|
1 2 3 4 5 6 7 8 9 10 |
Fonction saisie_atome():chaine Ecrire('donner un nom d une atome: ') Lire(atome) # Tant que le nom n’est pas valide, on redemande Tant que Non(verif_atome(atome)) faire Ecrire('donner un nom d une atome: ') Lire(atome) Fin tant que retourner atome Fin |
Déclaration des objets
| Objet | Type / Nature |
|---|---|
| atome | chaîne |
La procédure remplir_fichier_atomes
Le rôle de cette procédure est de créer et remplir le fichier Atomes.dat avec les informations de n atomes (nom et masse atomique).
1- La procédure remplir_fichier_atomes(n) ouvre le fichier Atomes.dat en mode écriture.
2- Elle répète l’opération n fois :
- saisit un nom d’atome valide grâce à la fonction saisie_atome(),
- saisit la masse atomique correspondante,
- écrit ces deux informations sur une seule ligne du fichier sous la forme : nom_atome masse_atomique
- Chaque atome est enregistré sur une ligne différente.
3- À la fin, le fichier est correctement fermé.
|
1 2 3 4 5 6 7 8 9 10 11 |
Procedure remplir_fichier_atomes(n:entier): Ecrire('*** Remplir le fichier Atomes.dat ***') Ouvrir(f , "Atomes.dat", "w") Pour i de 0 à n-1 faire atome <- saisie_atome() masse <- float(input('donner sa masse atomique: ')) Ecrire(f, Convch(atome) + ' ' + Convch(masse) + '\n') Fin pour Fermer(f) Fin |
Déclaration des objets
| Objet | Type / Nature |
|---|---|
| f | fichier |
| i | entier |
| atome | chaîne |
| masse | réel |
La fonction verif_formule
Le rôle de cette fonction est de vérifier si une chaîne de caractères représente une formule chimique valide, selon des règles simples de syntaxe.
1- La fonction commence par vérifier que la chaîne a au moins 2 caractères.
2- Elle teste ensuite :
-le premier caractère : doit être une lettre (A–Z ou a–z),
- le dernier caractère : doit être un chiffre (0–9).
3- Elle parcourt ensuite toute la chaîne pour vérifier que chaque caractère est soit : une lettre ou un chiffre.
4- Si toutes les conditions sont respectées, la fonction retourne Vrai, sinon Faux.
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 |
Fonction verif_formule(ch:chaine):booleen Si long(ch) >= 2 alors Si (('A'<=Majus(ch[0])<= 'Z') et ('0'<=ch[long(ch)-1]<='9')) alors i <- 0 Tant que (i<long(ch)-1) et (('A'<= Majus(ch[i])<='Z') ou ('0'<=ch[i]<= '9')) faire i <- i + 1 Fin tant que retourner (('A' <= Majus(ch[i]) <= 'Z') or ('0' <= ch[i] <= '9')) Sinon retourner Faux Fin si Sinon retourner Faux Fin si Fin |
Déclaration des objets
| Objet | Type / Nature |
|---|---|
| i | entier |
La fonction saisie_formule()
Le rôle de cette fonction est de permettre la saisie d’une formule chimique valide et garantir qu’elle respecte les règles de validation définies.
1- La fonction saisie_formule() demande à l’utilisateur d’entrer une formule chimique.
2- Elle utilise la fonction verif_formule(formule) pour vérifier la validité de la formule saisie.
3- Tant que la formule ne respecte pas les règles (formule invalide), la saisie est redemandée.
4- Une fois une formule correcte entrée, la fonction retourne cette formule.
|
1 2 3 4 5 6 7 8 9 10 |
Fonction saisie_formule():chaine Ecrire('donner la formule chimique de la molecule : ') Lire(formule) # Tant que le nom n’est pas valide, on redemande Tant que Non(verif_formule(formule)) faire Ecrire('donner la formule chimique de la molecule : ') Lire(formule) Fin tant que retourner formule Fin |
Déclaration des objets
| Objet | Type / Nature |
|---|---|
| formule | chaîne |
La procédure remplir_fichier_molecules
Le rôle de cette procédure est de créer et remplir le fichier Molecules.txt avec les noms des molécules et leurs formules chimiques valides.
1- La procédure remplir_fichier_molecules(m) ouvre le fichier Molecules.txt en mode écriture.
2- Elle répète l’opération m fois :
- saisit le nom de la molécule,
- saisit et valide sa formule chimique à l’aide de la fonction saisie_formule(),
- écrit ces informations dans le fichier selon le format : nom_molecule*formule
- Chaque molécule est enregistrée sur une ligne distincte.
3- À la fin, le fichier est correctement fermé.
|
1 2 3 4 5 6 7 8 9 10 11 |
Procedure remplir_fichier_molecules(m:entier) Ecrire('*** Remplir le fichier Molecule.txt ***') Ouvri(f , "Molecules.txt", "w") Pour i de 0 à m-1 faire Ecrire ('donner le nom de la molecule : ') Lire(molecule) formule <- saisie_formule() Ecrire(f , molecule + '*' + formule + '\n') Fin pour Fermer(f) Fin |
Déclaration des objets
| Objet | Type / Nature |
|---|---|
| f | fichier |
| i | entier |
| molecule | chaîne |
| formule | chaîne |
La procédure extraire_noms_nombres_atomes
Le rôle de cette procédure est d' analyser une formule chimique et en extraire : les noms des atomes,le nombre (indice) de chaque atome, puis stocker ces informations dans deux tableaux distincts.
1- La procédure extraire_noms_nombres_atomes(formule, noms_atomes, nombres_atomes) reçoit :
- une formule chimique (ex : "H2O1"),
- un tableau pour les noms des atomes,
- un tableau pour les nombres d’atomes.
2- Elle initialise les tableaux et remet à zéro les longueurs globales.
3- Elle parcourt la formule caractère par caractère :
- Les lettres sont regroupées pour former le nom d’un atome.
- Les chiffres sont regroupés pour former le nombre d’atomes correspondant.
- Chaque nom d’atome est stocké dans noms_atomes.
- Chaque nombre associé est stocké dans nombres_atomes.
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 |
Procedure extraire_noms_nombres_atomes(formulechaine, noms_atomes:tableau, nombres_atomes:tableau) # Initialisation des tableaux Pour i de 0 à longueur_noms-1 faire noms_atomes[i] <- '' nombres_atomes[i] <_ 0 Fin pour longueur_noms <- 0 longueur_nombres <- 0 mot <- "" nb <- "" Pour i de 0 à long(formule))-1 faire Si 'A' <= Majus(formule[i].upper()) <= 'Z' alors mot <- mot + formule[i] Si nb != "" alors nombres_atomes[longueur_nombres] = int(nb) longueur_nombres <- longueur_nombres + 1 nb <- "" Sinon si '0' <= formule[i] <= '9' alors nb <- nb + formule[i] Si mot != "" alors noms_atomes[longueur_noms] <- mot longueur_noms = longueur_noms + 1 mot <- "" Fin si Fin si Fin pour # Ajout du dernier nombre s’il existe Si nb != "" alors nombres_atomes[longueur_nombres] <- int(nb) longueur_nombres <- longueur_nombres + 1 Fin si Fin |
Déclaration des objets
| Objet | Type / Nature |
|---|---|
| i | entier |
| longueur_noms | entier (variable globale) |
| longueur_nombres | entier (variable globale) |
| mot | chaîne |
| nb | chaîne |
La fonction recherche_atome
Le rôle de cette fonction est de rechercher un atome donné dans un tableau de noms d’atomes et retourner son indice.
1- La fonction recherche_atome(atome, noms_atomes) parcourt le tableau noms_atomes.
2- Elle compare chaque élément du tableau avec le nom de l’atome recherché.
3- La recherche se fait séquentiellement depuis l’indice 0 jusqu’à longueur_noms − 1.
4- Dès que l’atome est trouvé, la boucle s’arrête. La fonction retourne l’indice.
|
1 2 3 4 5 6 7 |
Fonction recherche_atome(atome:chaine, noms_atomes:tableau):entier i <- 0 Tant que (i < longueur_noms) et (atome != noms_atomes[i]) faire i <- i + 1 Fin tant que retourner i Fin |
Déclaration des objets
| Objet | Type / Nature |
|---|---|
| i | entier |
| longueur_noms | entier (variable globale) |
La procédure remplir_fichier_resultats
Le rôle de cette procédure est de calculer la masse molaire de chaque molécule du fichier Molecules.txt et enregistrer les résultats dans Resultat.dat.
Explication détaillée du fonctionnement :
1- Ouverture des fichiers :
- Molecules.txt : contient les molécules et leurs formules (nom_molecule*formule).
- Resultat.dat : fichier où seront enregistrés les noms des molécules et leur masse molaire.
2- Lecture des molécules :
Chaque ligne du fichier Molecules.txt est lue et la formule chimique est extraite après le *.
3- Extraction des atomes et de leurs nombres :
La fonction extraire_noms_nombres_atomes décompose la formule en deux tableaux :
- noms_atomes : les symboles des atomes présents,
- nombres_atomes : le nombre d’atomes correspondant.
4- Calcul de la masse moléculaire :
Pour chaque atome, on lit son symbole et sa masse atomique depuis Atomes.dat.
On cherche l’indice de l’atome dans le tableau avec reherche_atome.
On multiplie la masse atomique par le nombre d’atomes dans la molécule et on additionne au total.
5- Écriture des résultats :
Le nom de la molécule et sa masse molaire calculée sont : affichés à l’écran et écrits dans Resultat.dat.
6- Fermeture des fichiers :
Tous les fichiers sont correctement fermés après traitement.
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 |
Procédure remplir_fichier_resultats(noms_atomes:tableau, nombres_atomes:tableau) Ecrire('*** Remplir le fichier Resultat.dat ***') Ouvrir(f_resultat , "Resultat.dat", "w") Ouvrir (f_molecules , open("Molecules.txt", "r") molecules <- Lire_lignes(f_molecules) Pour molecule dans molecules faire molecule <- molecule.strip('\n') formule <- Sous_chaine(molecule, Pos(moleculePos('*')+1,long(molecule) extraire_noms_nombres_atomes(formule, noms_atomes, nombres_atomes) masse <- 0 Ouvrir(f_atome , "Atomes.dat", "r") atome <- Lire_ligne(f_atome) Tant que (atome != "") faire atome <- atome.strip('\n') i <- reherche_atome(Sous_chaine(atome , 0 ,Pos(atome,' '), noms_atomes) masse = masse + Valeur(Sous_chaine(atome,Pos(atome, ' ') + 1, long(atome)) * nombres_atomes[i] atome <- Lire_ligne(f_atome) Fin tant que Ecrire(f_resultat, Sous_chaine(molecule, 0 , Pos(molecule,'*'))) + ', masse= ' + Convch(masse) + ' g/mol\n') Ecrire(Sous_chaine(molecule ,0, Pos(molecule,'*')), ', masse= ', masse, 'g/mol') Fermer(f_atome) Fin pour Fermer(f_resultat) Fermer(f_molecules) Fin |
Déclaration des objets
| Objet | Type / Nature |
|---|---|
| f_resultat | fichier |
| f_molecules | fichier |
| f_atome | fichier |
| molecules | chaîne |
| formule | chaîne |
| masse | réel |
| atome | chaîne |
| i | entier |
Solution en Python
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128 129 130 131 132 133 134 135 136 137 138 139 140 141 142 143 144 145 146 147 148 149 150 151 152 153 154 155 156 157 158 159 160 161 162 163 164 165 166 167 168 169 170 171 172 173 174 175 176 177 178 179 180 181 182 183 184 185 186 187 188 189 190 191 192 193 194 195 196 197 198 199 200 201 202 |
from numpy import * # -------------------------------------------------- # Déclaration de tableaux pour stocker : # - les noms des atomes # - le nombre d’atomes dans une molécule # -------------------------------------------------- noms_atomes = array([str] * 100) nombres_atomes = array([int()] * 100) # -------------------------------------------------- # Fonction qui permet de saisir un entier n # tel que : inf < n < sup # -------------------------------------------------- def saisie(inf, sup): # Première saisie de n n = int(input('donner n tel que ' + str(inf) + '< n < ' + str(sup) + ': ')) # Tant que n n’appartient pas à l’intervalle ]inf , sup[ # on redemande la saisie while (n <= inf) or (n >= sup): n = int(input('donner n tel que ' + str(inf) + '< n < ' + str(sup) + ': ')) # Retourne la valeur valide return n # -------------------------------------------------- # Fonction qui vérifie si une chaîne représente # un nom d’atome valide (lettres uniquement) # -------------------------------------------------- def verif_atome(ch): i = 0 # Parcours de la chaîne tant que ce sont des lettres while (i < len(ch) - 1) and ('A' <= ch[i].upper() <= 'Z'): i = i + 1 return ('A' <= ch[i].upper() <= 'Z') # -------------------------------------------------- # Fonction qui saisit et valide le nom d’un atome # -------------------------------------------------- def saisie_atome(): atome = input('donner un nom d une atome: ') # Tant que le nom n’est pas valide, on redemande while not verif_atome(atome): atome = input('donner un nom d une atome: ') return atome # -------------------------------------------------- # Procedure qui remplit le fichier Atomes.dat # Chaque ligne contient : nom_atome masse_atomique # -------------------------------------------------- def remplir_fichier_atomes(n): print('*** Remplir le fichier Atomes.dat ***') f = open("Atomes.dat", "w") for i in range(n): atome = saisie_atome() masse = float(input('donner sa masse atomique: ')) f.write(str(atome) + ' ' + str(masse) + '\n') f.close() # -------------------------------------------------- # Fonction qui vérifie la validité d’une formule chimique # (lettres et chiffres, commence par une lettre # et se termine par un chiffre) # -------------------------------------------------- def verif_formule(ch): if len(ch) >= 2: if (('A' <= ch[0].upper() <= 'Z') and ('0' <= ch[len(ch)-1] <= '9')): i = 0 while (i < len(ch) - 1) and (('A' <= ch[i].upper() <= 'Z') or ('0' <= ch[i] <= '9')): i = i + 1 return (('A' <= ch[i].upper() <= 'Z') or ('0' <= ch[i] <= '9')) else: return False else: return False # -------------------------------------------------- # Fonction qui saisit et valide une formule chimique # -------------------------------------------------- def saisie_formule(): formule = input('donner la formule chimique de la molecule : ') while verif_formule(formule) == False: formule = input('donner la formule chimique de la molecule : ') return formule # -------------------------------------------------- # Procedure qui remplit le fichier Molecules.txt # Format : nom_molecule*formule # -------------------------------------------------- def remplir_fichier_molecules(m): print('*** Remplir le fichier Molecule.txt ***') f = open("Molecules.txt", "w") for i in range(m): molecule = input('donner le nom de la molecule : ') formule = saisie_formule() f.write(molecule + '*' + formule + '\n') f.close() # -------------------------------------------------- # Procedure qui extrait : # - les noms des atomes # - le nombre de chaque atome # à partir d’une formule chimique # -------------------------------------------------- def extraire_noms_nombres_atomes(formule, noms_atomes, nombres_atomes): global longueur_noms, longueur_nombres # Initialisation des tableaux for i in range(longueur_noms): noms_atomes[i] = '' nombres_atomes[i] = 0 longueur_noms = 0 longueur_nombres = 0 mot = "" nb = "" for i in range(len(formule)): if 'A' <= formule[i].upper() <= 'Z': mot = mot + formule[i] if nb != "": nombres_atomes[longueur_nombres] = int(nb) longueur_nombres += 1 nb = "" elif '0' <= formule[i] <= '9': nb = nb + formule[i] if mot != "": noms_atomes[longueur_noms] = mot longueur_noms += 1 mot = "" # Ajout du dernier nombre s’il existe if nb != "": nombres_atomes[longueur_nombres] = int(nb) longueur_nombres += 1 # -------------------------------------------------- # Fonction qui recherche un atome dans le tableau # et retourne son indice # -------------------------------------------------- def reherche_atome(atome, noms_atomes): global longueur_noms i = 0 while (i < longueur_noms) and (atome != noms_atomes[i]): i = i + 1 return i # -------------------------------------------------- # Procedure qui calcule la masse moléculaire # et remplit le fichier Resultat.dat # -------------------------------------------------- def remplir_fichier_resultats(noms_atomes, nombres_atomes): global longueur_noms, longueur_nombres print('*** Remplir le fichier Resultat.dat ***') f_resultat = open("Resultat.dat", "w") f_molecules = open("Molecules.txt", "r") molecules = f_molecules.readlines() for molecule in molecules: molecule = molecule.strip('\n') formule = molecule[molecule.find('*') + 1:] extraire_noms_nombres_atomes(formule, noms_atomes, nombres_atomes) masse = 0 f_atome = open("Atomes.dat", "r") atome = f_atome.readline() while atome != "": atome = atome.strip('\n') i = reherche_atome(atome[:atome.find(' ')], noms_atomes) masse += float(atome[atome.find(' ') + 1:]) * nombres_atomes[i] atome = f_atome.readline() f_resultat.write(molecule[:molecule.find('*')] + ', masse= ' + str(masse) + ' g/mol\n') print(molecule[:molecule.find('*')], ', masse= ', masse, 'g/mol') f_atome.close() f_resultat.close() f_molecules.close() # -------------------------------------------------- # Programme principal # -------------------------------------------------- longueur_noms = 0 longueur_nombres = 0 n = saisie(0, 51) remplir_fichier_atomes(n) m = saisie(0, 51) remplir_fichier_molecules(m) remplir_fichier_resultats(noms_atomes, nombres_atomes) |
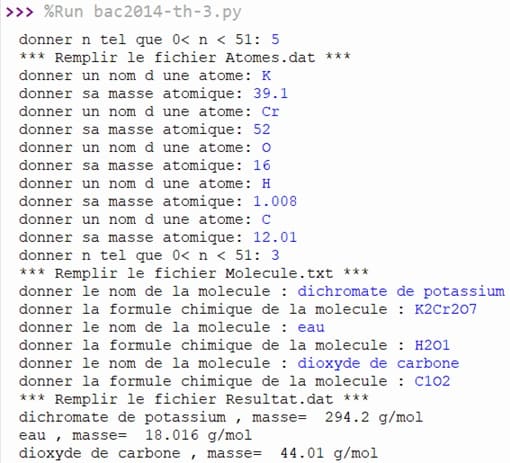
Exécution du programme
0 commentaire
laisser un commentaire
Tutoriels récents
Tutoriels populaires

Tutoriels plus commentés